# 目录
1.Module 1 - Introduction to RNA sequencing
2.Module 2 - RNA-seq Alignment and Visualization
3.Module 3 - Expression and Differential Expression
4.Module 4 - Isoform Discovery and Alternative Expression
- Reference Guided Transcript Assembly
- de novo Transcript Assembly
- Transcript Assembly Merge
- Differential Splicing
- Splicing Visualization
5.Module 5 - De novo transcript reconstruction
6.Module 6 - Functional Annotation of Transcripts
# 2.3 IGV
# 1.introduction
# Description of the lab
高通量测序最受欢迎的工具 -IGV(Integrative Genomics Viewer)
伴随本教程的文件
- IGV Lecture - Brief
- IGV Lecture - Long, from Broad Institute
完成本次教程可实现以下工作
可视化各种基因组数据
快速导航基因组
可视化 reads 比对情况
肉眼验证 SNP/SNV
# Requirements
Integrative Genomics Viewer
Ability to run Java
Note that while most tutorials in this course are performed on the cloud, IGV will always be run on your local machine
# Compatibility
本教程是为 IGV v2.3 准备的,可以在 IGV 下载页面上找到。强烈建议使用这个版本。
# Data Set for IGV
使用公开的来自 HCC1143 细胞系的 Illumina 序列数据。HCC1143 细胞系是从一名患有乳腺癌的 52 岁白人妇女体内产生的。这个细胞系的附加信息可以在这里找到:HCC1143 (tumor, TNM stage IIA, grade 3, primary ductal carcinoma) 以及 HCC1143/BL(matched normal EBV transformed lymphoblast cell line).
从细胞系 HCC1143 产生的 reads 比对到这个区域
Chromosome 21: 19,000,000-20,000,000
HCC1143.normal.21.19M-20M.bam
HCC1143.normal.21.19M-20M.bam.bai
# 2. Getting familiar with IGV
# Get familiar with the interface
载入一个基因组:
默认情况下,IGV 加载 Human hg19。如果你研究的是另一个版本的人类基因组,或者另一种物种,你可以通过点击左上角的下拉菜单来改变基因组。在这个教程中,我们将使用人类 hg19。
也可以采用以下方式 ( File -> Load from Server... ):
- Ensembl genes (or your favourite source of gene annotations)
- GC Percentage
- dbSNP 1.3.1 or 1.3.7
Navigation:
在这个参考基因组中可以看到染色体列表,选择 1 号染色体。
location 字段 (在界面的左上角) 中输入,导航到 chr1:10 000- 11000,然后单击 Go。这显示了 1 号染色体的窗口宽 1000 个碱基对,从 10000 号位置开始。
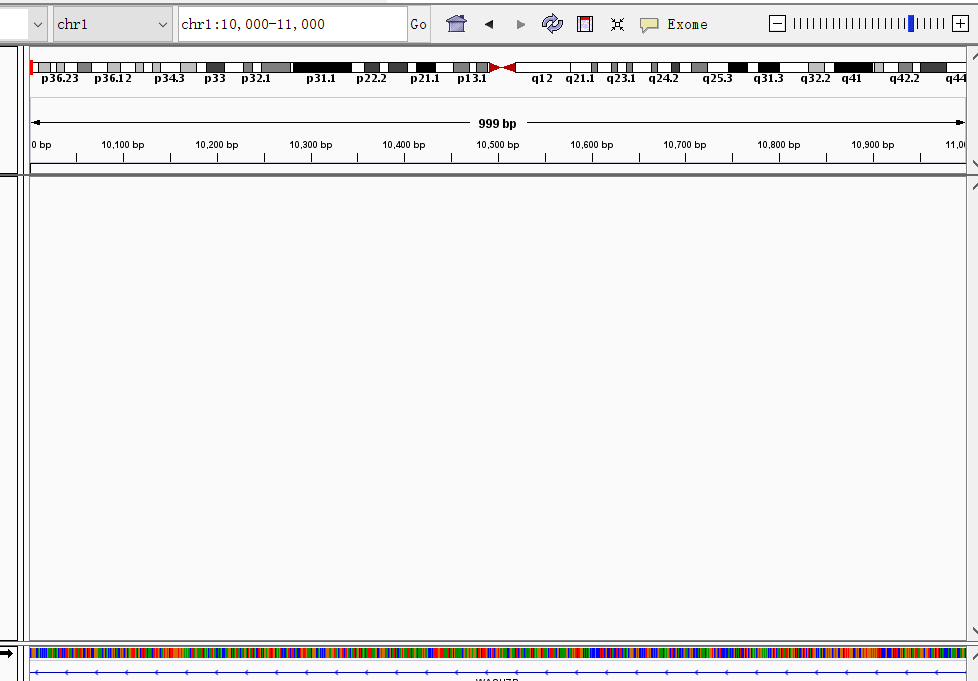
IGV 以颜色序列的形式显示基因组中的碱基序列 (例如 A = 绿色,C = 蓝色,等等)。这使得重复序列,比如在这个区域开始处发现的那些序列,很容易识别。放大一点使用 + 按钮看到参考基因组序列的单个碱基。
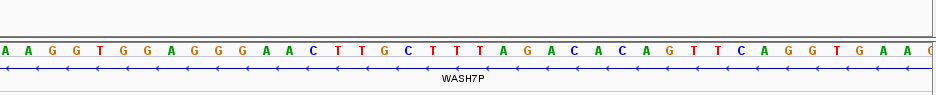
你可以在基因组坐标所在的框中输入你感兴趣的基因,然后按 Enter/Return 键。试试你最喜欢的基因,或者 BRCA1。
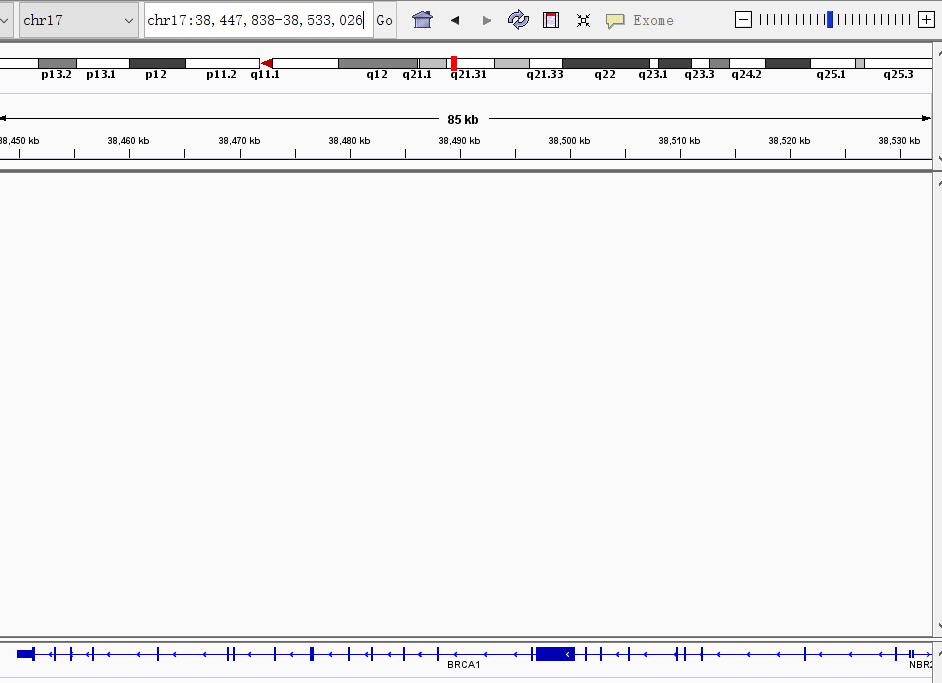
基因用线和框表示。线代表内含子区域,框代表外显子区域。箭头表示该基因的转录方向 / 链。当一个外显子框变窄,这表示一个 UTR。
# Region Lists
有时,保存当前位置或加载感兴趣的区域真的很有用。为此,IGV 中有一个区域导航器。要访问它,单击 Regions > Region Navigator。在浏览基因组时,可以随时按 Add 按钮保存一些书签。
# Loading Read Alignments
我们将使用乳腺癌细胞系 HCC1143 来可视化比对结果。在速度方面,只有一小部分 chr21 将装载 (19M:20M)。
HCC1143 Alignments to hg19:
- HCC1143.normal.21.19M-20M.bam
- HCC1143.normal.21.19M-20M.bam.bai
复制文件到你的本地,并在 IGV 中选择 File > Load from File... ,选择 bam 文件,并单击 OK。注意,为了让 IGV 正确地加载它们,bam 文件和索引文件必须在同一个目录中。
# Visualizing read alignments
选择染色体位点: chr21:19,480,041-19,480,386
To start our exploration, right click on the track-name, and select the following options:
- Sort alignments by
start location - Group alignments by
pair orientation
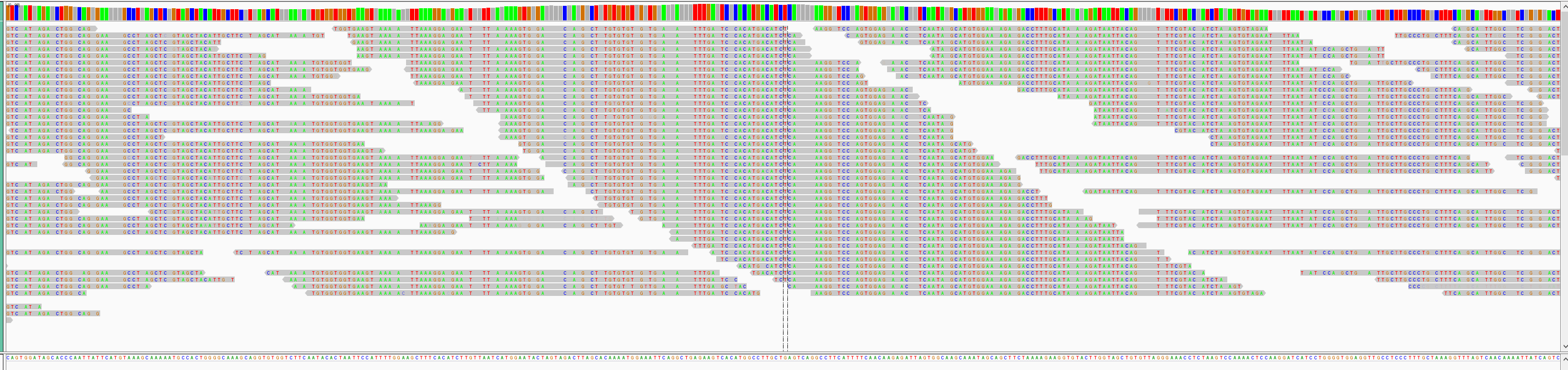
通过右键点击比对界面和切换选项来试验各种设置。想想哪一种方法最适合特定的任务 (例如,质量控制、SNP 调用、CNV 查找)。
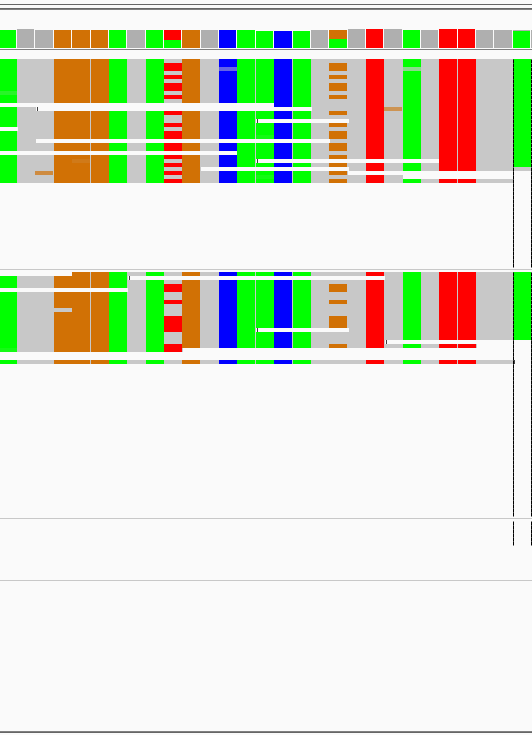
# 3.Inspecting SNPs, SNVs, and SVs
# Two neighbouring SNPs
- Navigate to region
chr21:19,479,237-19,479,814 - Note two heterozygous variants, one corresponds to a known dbSNP (
G/Ton the right) the other does not (C/Ton the left) - Zoom in and center on the
C/TSNV on the left, sort by base (windowchr21:19,479,321is the SNV position) - Sort alignments by
base - Color alignments by
read strand

# Homopolymer region with indel
Navigate to position chr21:19,518,412-19,518,497

# Coverage by GC
Navigate to position chr21:19,611,925-19,631,555 . Note that the range contains areas where coverage drops to zero in a few places.
**Example **
- Use
Collapsedview - Use
Color alignments by->insert size and pair orientation - Load GC track
- See concordance of coverage with GC content
# Heterozygous SNPs on different alleles
Navigate to region chr21:19,666,833-19,667,007
**Example **
- Sort by base (at position
chr21:19,666,901)
对于这两个 snp,等位基因之间没有联系,因为两个 snp 的 reads 都只包含一个或另一个
# 4.Automating Tasks in IGV
我们可以使用 Tools 菜单调用运行批处理脚本。IGV 网站描述了批处理脚本:
Batch file requirements: https://www.broadinstitute.org/igv/batch
Commands recognized in a batch script: https://www.broadinstitute.org/software/igv/PortCommands
We also need to provide sample attribute file as described here: http://www.broadinstitute.org/software/igv/?q=SampleInformation
下载数据集的批处理脚本和属性文件:
- Batch script: Run_batch_IGV_snapshots.txt
- Attribute file: Igv_HCC1143_attributes.txt