# 将 SRA 转为 fastq
数据下载好之后,我们得到的是 SRR 文件,需要将其转换为 fastq 格式才能使用
wkd=/home/project/single-cell/MCC | |
cat SRR_Acc_List-2586-4.txt |while read i | |
do | |
fastq-dump --gzip --split-files ${i}.sra | |
done |
结束之后每个 SRR 文件会变成解压出三个文件
为什么会有三个文件,这个需要去查一下 10X 的官方说明
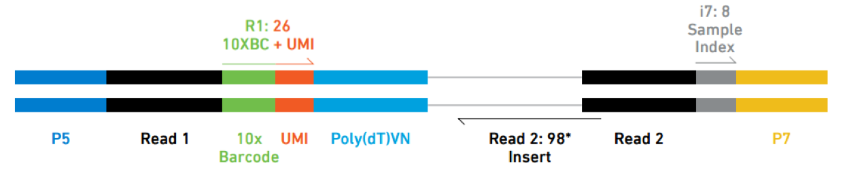
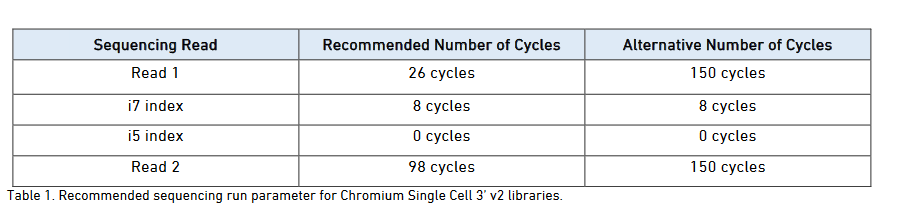
可以看到 Read1 是 26bp,主要是 16bp 的 barcode 和 10bp 的 UMI,Read2 的长度不固定,所以不一定是 98bp,视情况而定,还有一个 I7 index 长度是 8bp
接下来就按照 10X 的标准去对文件进行改名
cat SRR_Acc_List-9245-3.txt | while read i | |
do | |
mv ${i}_1*.gz${i}_S1_L001_I1_001.fastq.gz | |
mv ${i}_2*.gz ${i}_S1_L001_R1_001.fastq.gz | |
mv ${i}_3*.gz ${i}_S1_L001_R2_001.fastq.gz | |
done |
之后我创建了文件夹将每个 SRR 数据的三个 fastq 文件放到一个文件夹下
SRR7722937 | |
├── SRR7722937_S1_L001_I1_001.fastq.gz | |
├── SRR7722937_S1_L001_R1_001.fastq.gz | |
└── SRR7722937_S1_L001_R2_001.fastq.gz |
# cellranger 使用
下载 cellranger3.1 版本(目前 cellranger 已经支持 4.0 版本)并添加至环境变量
curl -o cellranger-3.1.0.tar.gz "https://cf.10xgenomics.com/releases/cell-exp/cellranger-3.1.0.tar.gz?Expires=1601922176&Policy=eyJTdGF0ZW1lbnQiOlt7IlJlc291cmNlIjoiaHR0cHM6Ly9jZi4xMHhnZW5vbWljcy5jb20vcmVsZWFzZXMvY2VsbC1leHAvY2VsbHJhbmdlci0zLjEuMC50YXIuZ3oiLCJDb25kaXRpb24iOnsiRGF0ZUxlc3NUaGFuIjp7IkFXUzpFcG9jaFRpbWUiOjE2MDE5MjIxNzZ9fX1dfQ__&Signature=mCC-emSQTqWg3i6Rm23lkuccNRI4z7xf-8bJ-O5gHqsYFJHDPZN9MeUZW5vhRZvmZhiGK7EDL4y~3xPtQbS6JtfUL9EFsoUxMywyF7tGN2ZlU2pbb2EVpmhKjmDWPVmvurDb~ZlHVYZCYcOc6gEHVtFRre-ICa7-nccVtnUJA-HvxNrZMs5mlQaUG9E-ngtbLi86gvIYlMHYnFRieZYpMA-kmUKrHGG8MhiuBGR96AWOrsVdTyMjD-BJOurGYvZGddWAF5uVXNPJs47FqF4fNCSQw71WOSRx4bQGdfb-jrOFp-NDSYFhkY1-gigku8VCx9phFCtyTAVM9yHeGN1oMQ__&Key-Pair-Id=APKAI7S6A5RYOXBWRPDA" | |
tar -zxvf cellranger-3.1.0.tar.gz | |
export PATH=/datadisk02/ScRNAseq/cellranger-3.1.0:$PATH |
构建注释
wget ftp://ftp.ensembl.org/pub/release-93/fasta/homo_sapiens/dna/Homo_sapiens.GRCh38.dna.primary_assembly.fa.gz | |
gunzip Homo_sapiens.GRCh38.dna.primary_assembly.fa.gz | |
wget ftp://ftp.ensembl.org/pub/release-93/gtf/homo_sapiens/Homo_sapiens.GRCh38.93.gtf.gz | |
gunzip Homo_sapiens.GRCh38.93.gtf.gz | |
cellranger mkgtf Homo_sapiens.GRCh38.93.gtf Homo_sapiens.GRCh38.93.filtered.gtf \ | |
--attribute=gene_biotype:protein_coding \ | |
--attribute=gene_biotype:lincRNA \ | |
--attribute=gene_biotype:antisense \ | |
--attribute=gene_biotype:IG_LV_gene \ | |
--attribute=gene_biotype:IG_V_gene \ | |
--attribute=gene_biotype:IG_V_pseudogene \ | |
--attribute=gene_biotype:IG_D_gene \ | |
--attribute=gene_biotype:IG_J_gene \ | |
--attribute=gene_biotype:IG_J_pseudogene \ | |
--attribute=gene_biotype:IG_C_gene \ | |
--attribute=gene_biotype:IG_C_pseudogene \ | |
--attribute=gene_biotype:TR_V_gene \ | |
--attribute=gene_biotype:TR_V_pseudogene \ | |
--attribute=gene_biotype:TR_D_gene \ | |
--attribute=gene_biotype:TR_J_gene \ | |
--attribute=gene_biotype:TR_J_pseudogene \ | |
--attribute=gene_biotype:TR_C_gene | |
cellranger mkref --genome=GRCh38 \ | |
--fasta=Homo_sapiens.GRCh38.dna.primary_assembly.fa \ | |
--genes=Homo_sapiens.GRCh38.93.filtered.gtf \ | |
--ref-version=3.0.0 |
也可以下载构建好的注释
curl -O https://cf.10xgenomics.com/supp/cell-exp/refdata-cellranger-GRCh38-3.0.0.tar.gz |
然后进行比对即可
cellranger count --id=SRR937 --transcriptome=GRCh38 --fastqs=SRR7722937/ --sample=SRR7722937 |
在分析过程中可以发现有些命令比较眼熟,不难发现 cellranger 的比对还是构建索引其实都有 STAR 的影子,后续的话我将 STARsolo(利用 STAR 分析单细胞数据)的流程再整理一下。
Generating STAR genome index (may take over 8 core hours for a 3Gb genome)... | |
15:12:38 ..... Started STAR run | |
15:12:38 ... Starting to generate Genome files | |
15:14:31 ... starting to sort Suffix Array. This may take a long time... | |
15:14:41 ... sorting Suffix Array chunks and saving them to disk... |
# 参考链接
https://support.10xgenomics.com/single-cell-gene-expression/software/release-notes/build#grch38_3.0.0
10x_Technical_Note_Base_Composition_SC3_v2_RevB.pdf
【生信技能树】使用 10X 单细胞转录组数据探索免疫治疗
单细胞实战 (三) Cell Ranger 使用初探
单细胞实战 (二) cell ranger 使用前注意事项