WGCNA 笔记第二弹
# 1.WGCNA 应用场景
不同组织样品
时间序列样品:生长发育,胁迫处理
单个材料:相同处理不同时间,不同处理相同时间,不同处理不同时间
两个材料:相同处理不同时间,不同处理相同时间,不同处理不同时间
表型数据:同级相关表型的数据
![image-20200801151521019]()
# 2.WGCNA 实验设计
1. 时间序列样品,两个材料相同处理,可以一起做 WGCNA 分析,也可以分别做然后比较,相同材料不同处理之间也一样
2. 同一物种,不同来源的转录组数据,可以放在一起做 WGCNA,也可以分开来比较
3. 一般建议 15 个样品以上进行 WGCNA 分析有生物学意义,可以使 5 个时间点三个重复的 15 个样品
4. 表型数据,建议收集可以统计量化的性状数据,可将模块和表型数据关联分析,有助于筛选关键基因模块和基因来解释相关表型
# 3. 单个材料案例
Floral Transcriptomes in Woodland Strawberry Uncover Developing Receptacle and Anther Gene Networks
野草莓花器官转录组解析花发育中花托和花药的基因调控网络
# 3.1 方案设计
单个材料不同组织样品
取材野草莓花器官不同组织
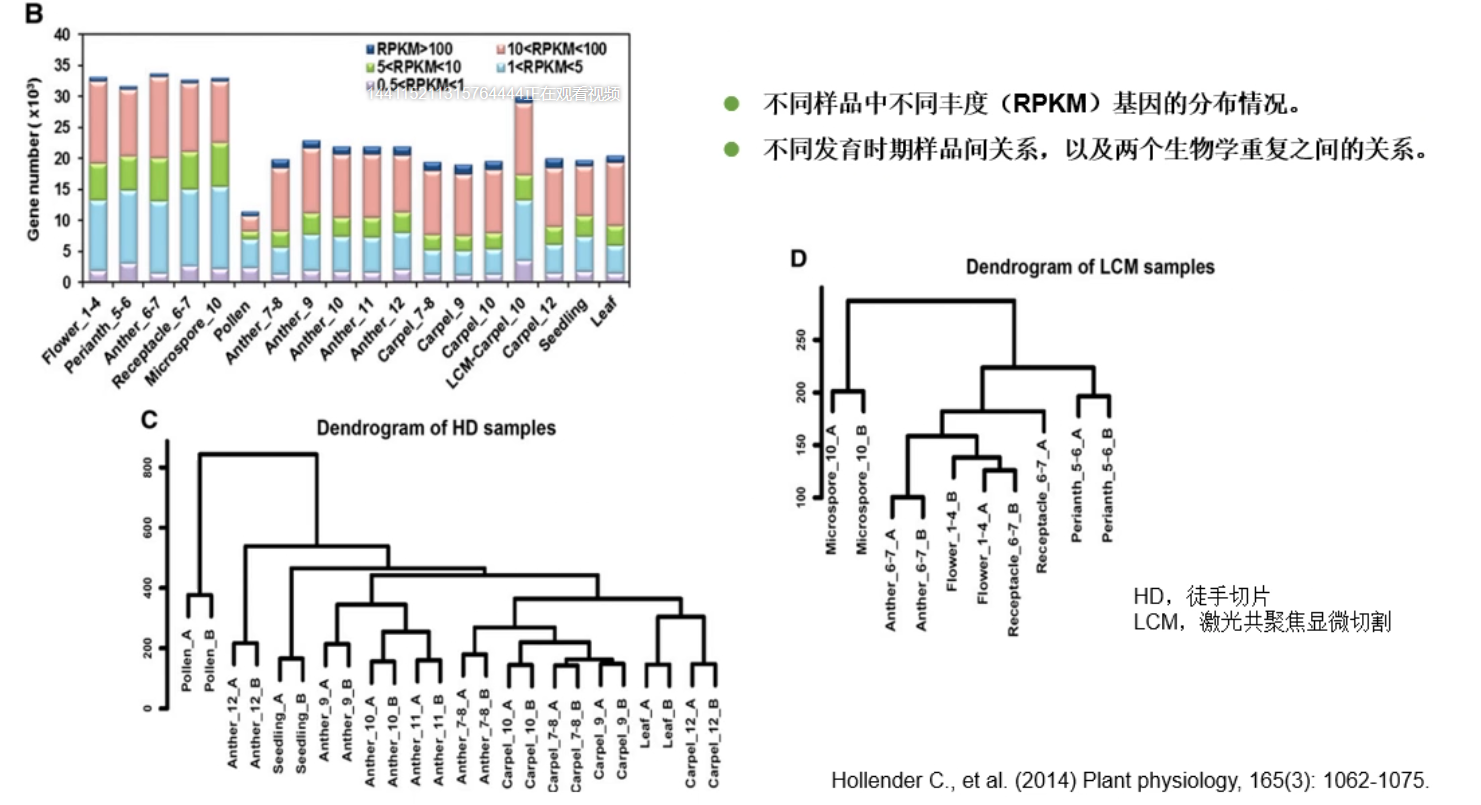
从不同发育时期的花器官分离花药、花粉、心皮、花托等组织,再加上小苗叶片,共 17 个组织。每个组织两个重复,共 34 个样本。
# 3.2 转录组数据整体分析
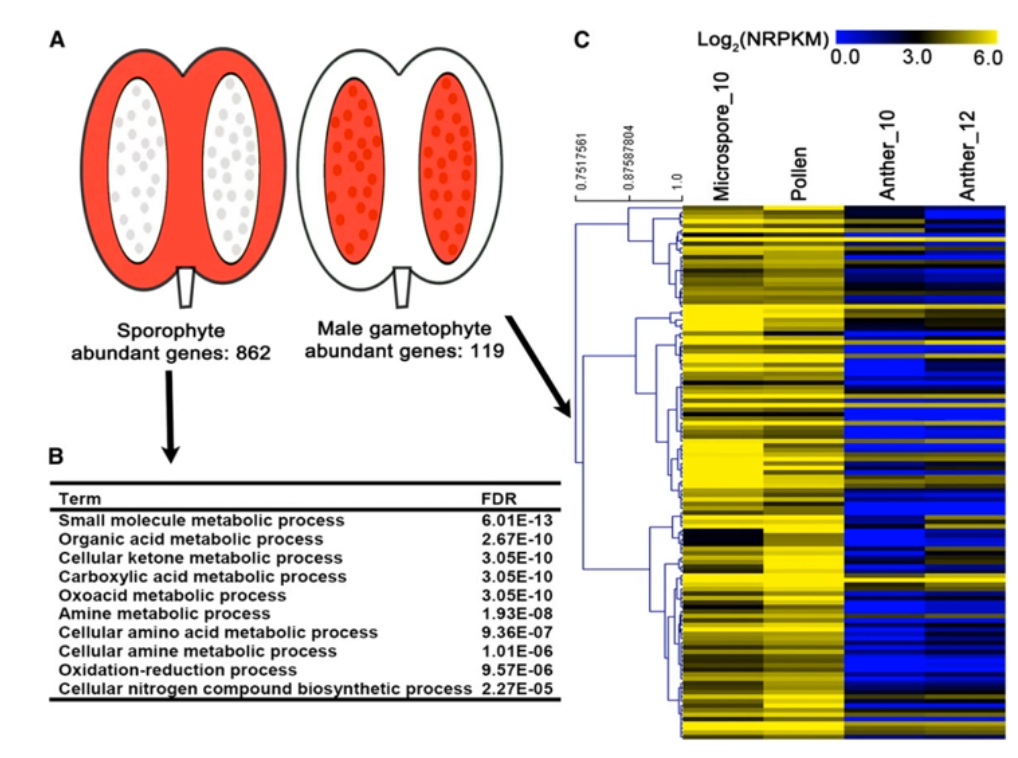
# 3.2 鉴定配子体孢子体相关基因
在不同发育时期花药中共同表达的基因鉴定为雄性孢子体特异性基因
GO 富集显示,雄性孢子基因集主要为代谢生物学过程,如小分子、有机酸、细胞内酮代谢等等
在花粉和小孢子特异表达的基因中共同表达的基因鉴定为配子体特异基因
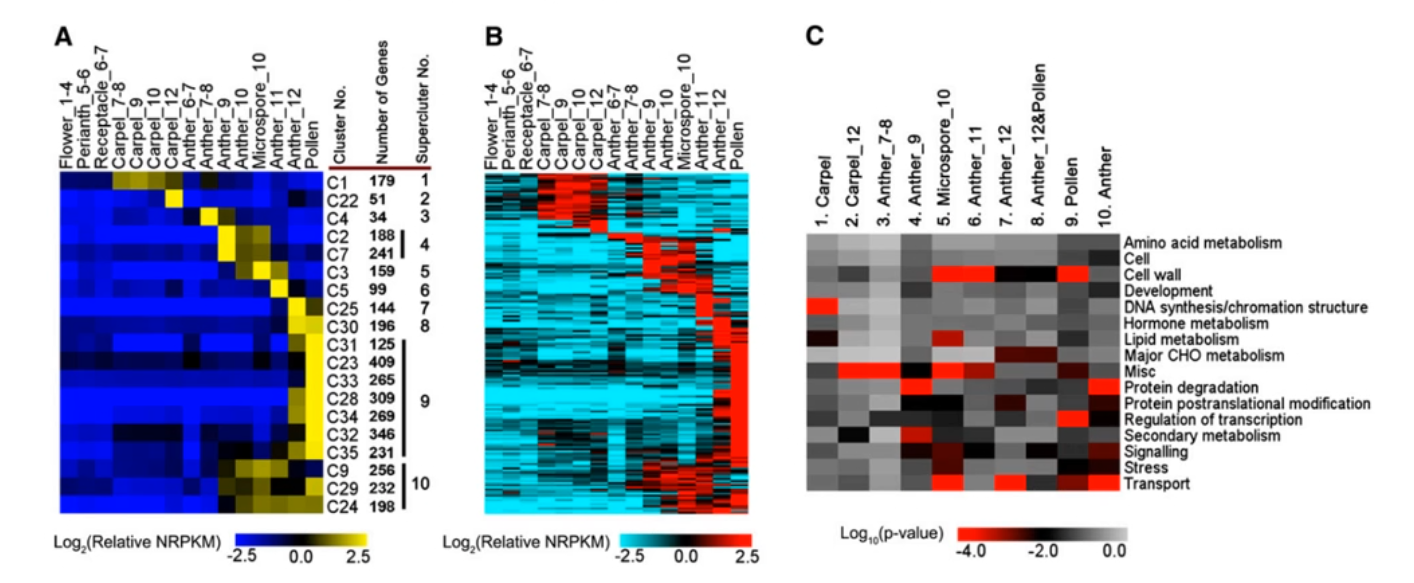
# 3.3 花发育过程转录组的时空表达模式分析
对所有差异表达的基因进行 K-means 聚类分析鉴定 19 个 cluster,呈现出发育时期和组织特异性表达模式,如 C1 C22 为心皮特异性
进一步将 19 个 cluster 合并为 10 个 supercluster,并进行功能富集分析,如心皮特异性的 supercluster,并进行功能富集分析。如心皮特异性的 supercluster1 富集的功能为 DNA 合成。
对 cluster 中的 161 个转录因子进行表达量聚类分析,也呈现发育时期和组织特异性的表达模式。
# 3.4 WGCNA 构建共表达网络
用差异表达基因进行 WGCNA 分析,鉴定了 23 个基因模块。
用每个模块的 epigengene 值与不同组织样品进行关联分析,鉴定组织紧密关联的基因模块。
12 个模块与单一的组织样品特异性高度相关,如 blue 模块与花粉(pollen)特异性相关。
WGCNA 鉴定的组织特异性基因集合与 K-means 的结果相符合
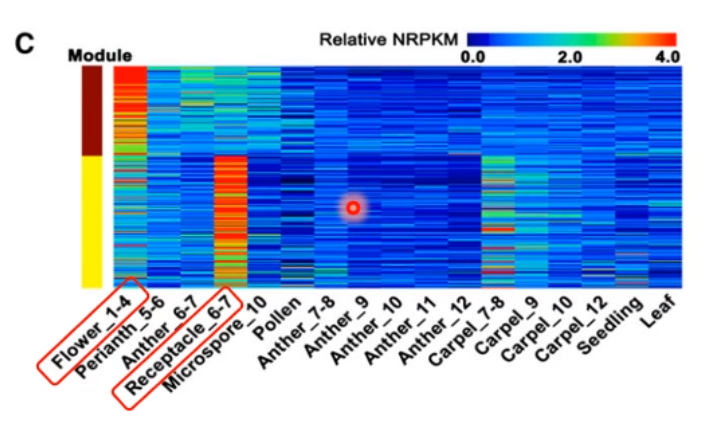
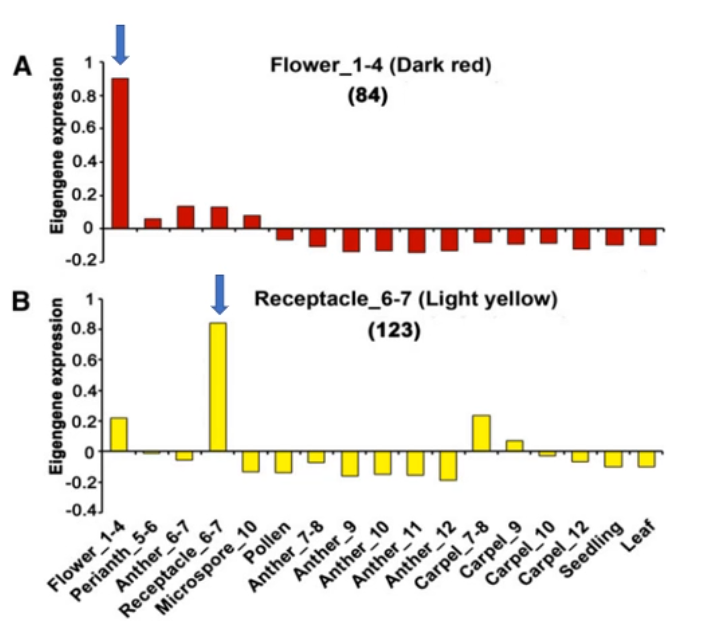
WGCNA 鉴定的花托特异性模块(light yellow)和幼嫩花(stage1-4)特异性模块(dark red),在 K-means 的 cluster 中并没有

# 3.5 组织特异性表达模块
WGCNA 鉴定的花托特异性模块(light yellow)和幼嫩花(stage1-4)特异性模块,在 K-means 的 cluster 中并没有。
分析了 Dark red 模块和 Light yellow 模块 epigengene 在各样品中的表达模式
热图呈现了 Dark red 模块和 Light yellow 模块中每个基因在各个样品中的表达模式
# 3.6 花托特异性模块内部分析
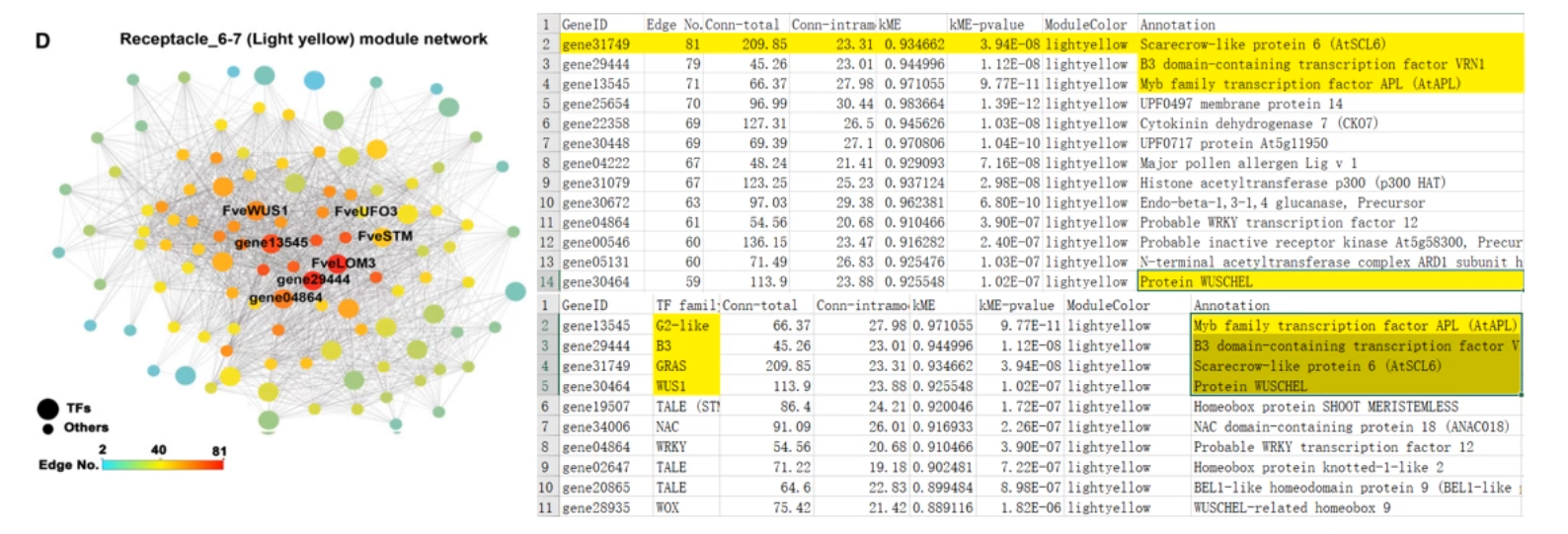
关键基因(hub gene):模块内网络中连接度较多的基因。
关注模块中的转录因子:花托特异性模块 111 个基因中有 27 个转录因子,重点分析这些转录因子。
大部分 hub gene 为参与调控分生组织的转录因子,如 WOX、GRS、NAC 等。
hub gene 中连接度最高的为 GRS 转录因子家族的 FveLOM3。拟南芥中三突变体 lom1 lom2 lom3 表现出分生组织异常的表型。因此 FveLOM3 可能是花托发育的关键调控因子。
此外,高连接度的 hub gene 还包括一个 B3 domain 转录因子、一个 Myb 转录因子和 WUS 同源基因
# 3.7 花药发育差异基因分析
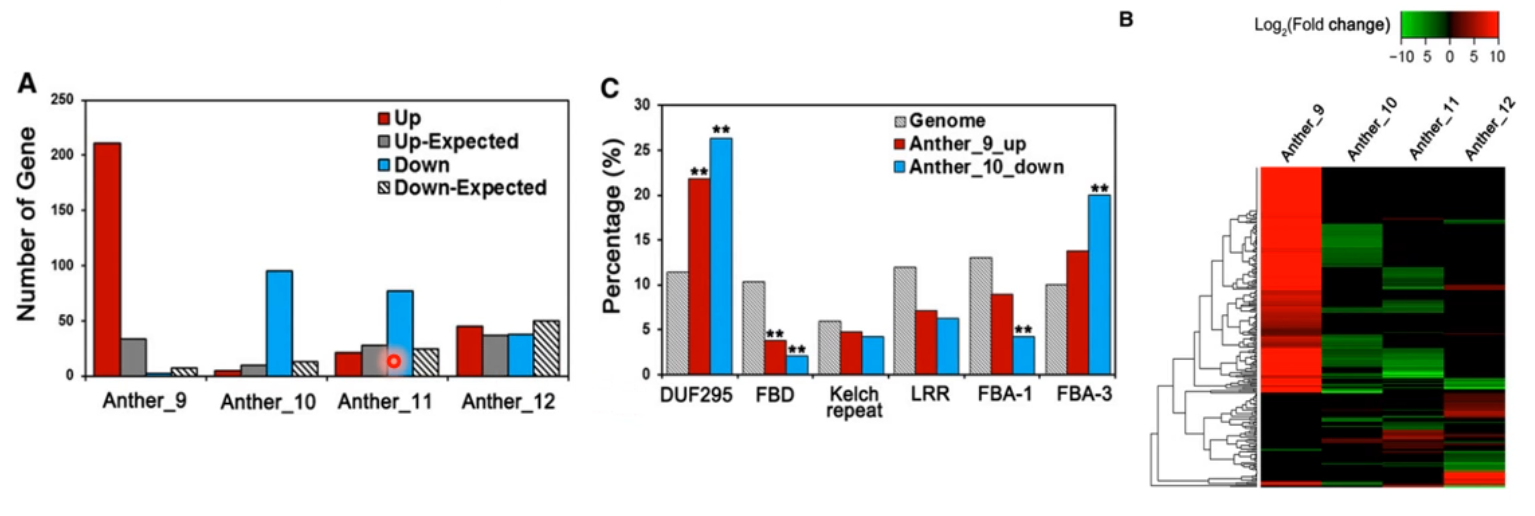
stage9 花药中上调表达的 1453 个基因中,211 个编码 FBX domain 蛋白。在野草莓基因组中包含 820 个 FBX 基因。大比例 FBX 上调表达说明在花药减数分裂这个时期发生了大量的蛋白降解。
K-means 的 cluster 富集分析中花药 - 9 的 cluster 富集的为蛋白降解。
FBX 基因表达聚类热图分析显示,大部分 FBX 只在 stage9 时期暂时高表达,随后的 stage10 和 11 下调表达。
在花药发育过程中,共 296 个 FBX 基因差异表达,其中 6 个亚家族占的比例较大,包括 FBD、LRR、DUF295
# 3.8 花药特异性模块分析
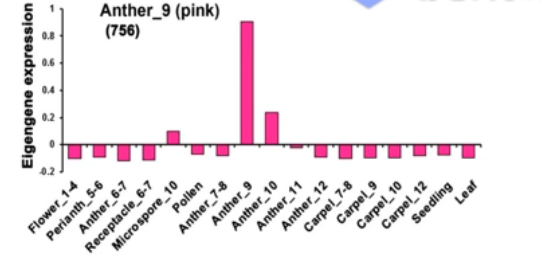
花药 stage9 特异性基因模块为 pink,其中包含了 37 个 FBX 基因。
通过筛选连接度较高的基因来鉴定 hub gene。5 个基因的度数目高于 200。
其中 4 个基因参与蛋白降解,F-box、WD-repeat

# 3.8 小结:WGCNA 分析思路
实验方案:单个材料,不同组织样品,所有差异表达基因进行 WGCNA 分析
通过模块 Epigengene 值与不同组织样本进行关联分析,鉴定组织特异性模块
hub 基因筛选
连接度较高的基因
重点关注转录因子(花托特异性模块)
前期结果中的目标基因(话要特异性模块中的 FBX)
# 4. 两个材料
Global transcriptome and coexpression network analyses reveal cultivara specific molecular signatures associated with seed development and seed size/weight determination in chickpea
# 4.1 方案设计
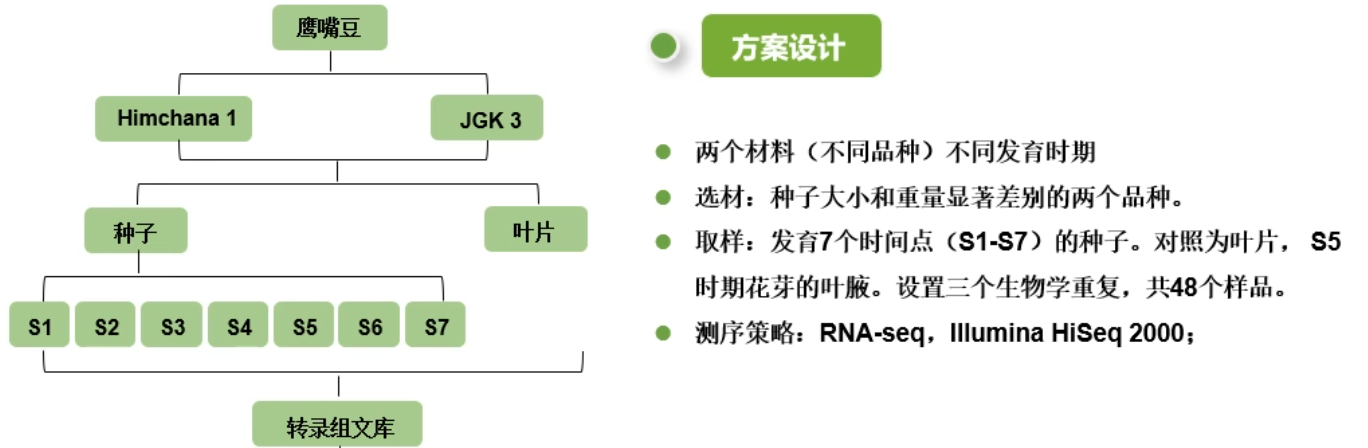
# 4.2 两个鹰嘴豆栽培种的种子发育表性分析
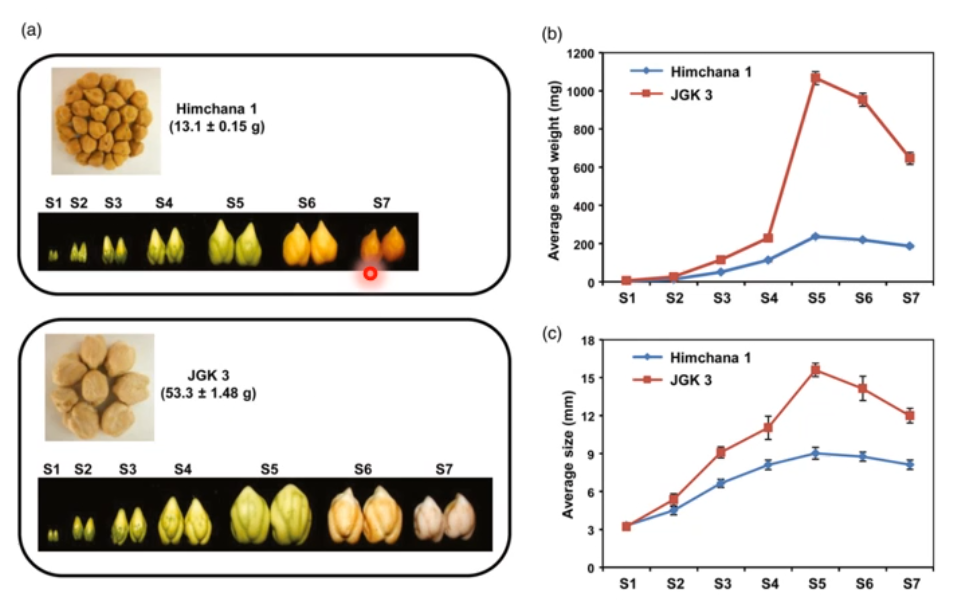
两个鹰嘴豆栽培种 Himchana1 (小种子,平均 100 粒种子重量为 13.1g) 和 JGK3 (大
种子,平均 100 粒种子重量为 53.3g).
种子发育的 7 个时间段(S1-S7), 分别代表了种子发育的三个阶段:胚芽发育(S1-S3)、早期和中期成熟阶段(籽粒灌浆,S4-S5)、成熟晚期(种子干燥,S6-S7)
种子的不同发育阶段 S1-S7 依据授粉天数(Day after Pollination,DAP) 划分,5、9.12,19,25、30 和 40 DAP 分别为 S1、S2、S3.S4、S5、S6 和 S7.
表型分析:种子发育不同时间点的种子重量和大小的统计数据比较。
# 4.3 两个材料种子发育过程转录组的整体解析
为了分析两个材料种子发育过程转录组动态变化的差异,基于 16 个组织样品所有表达基因的表达量的斯皮尔曼相关系数(SCC)进行层次聚类和主成分分析(PCA)。
两个材料中相同发育时期组织样品表现出很高的相关性。
两个材料叶片聚在一起,与所有种子样品表现出明显差异。
两个材料的 S3 有差异,JGK3-S3 与 S2 更接近,而 HC1-S3 与 S4 更像。这说明 HC1 在种子发育早期比 JGK3 生长发育得更快。
虽然两个材料 S5 也聚类在一起,但是关系并没有其他时期的紧密,也呈现一定的差异。
因此,S3 和 S5 可能是两个材料种子大小和重量差异的关键发育时期

# 4.4 种子发育过程中差异基因表达分析
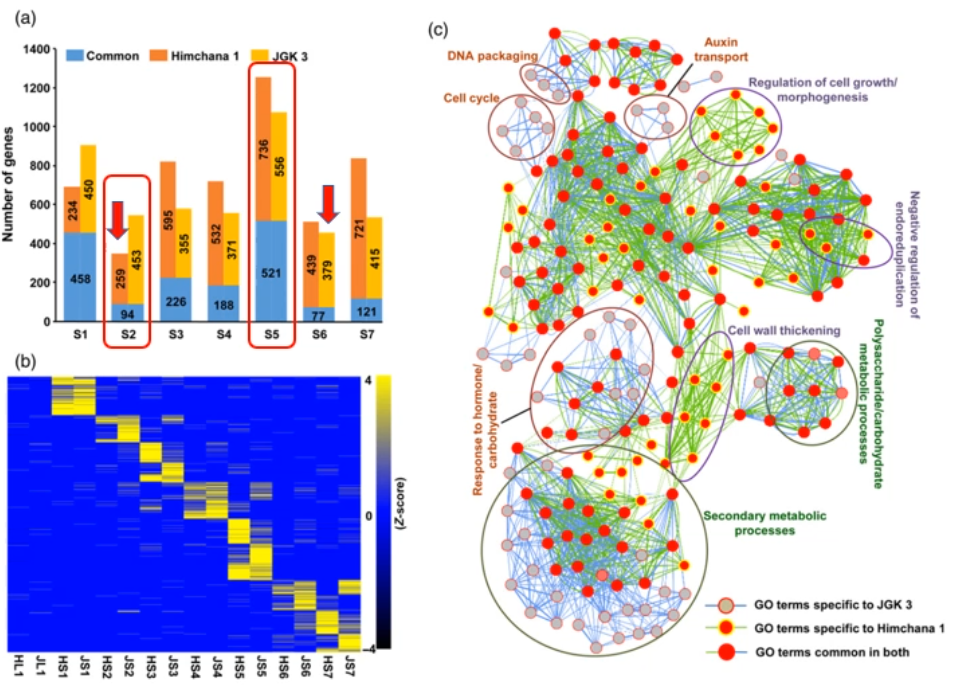
鉴定种子发育过程中每个时期特异表达的基因
各个时期特异表达的基因数目差异很大,S2 最少,S5 最多。
两个材料中各自时期特异表达的基因数目也有所不同,HC1 在 S2 最少,JGK3 则在 S6 最少,但是两者在 S5 都是最多的。
两个材料中均时期特异表达的基因比例也不小,表达量层次聚类分析呈现出明显的发育时期特异性。
说明每个发育阶段有着自己独立的发育程序。
Go 富集分析,主要为生殖过程、细胞壁组装、细胞周期和细胞分裂、碳代谢等,这些都是已知参与种子发育的。
有些 GOterm 在两个材料中均富集,有些只在一个材料中富集。
# 4.5 两个材料差异表达基因分析
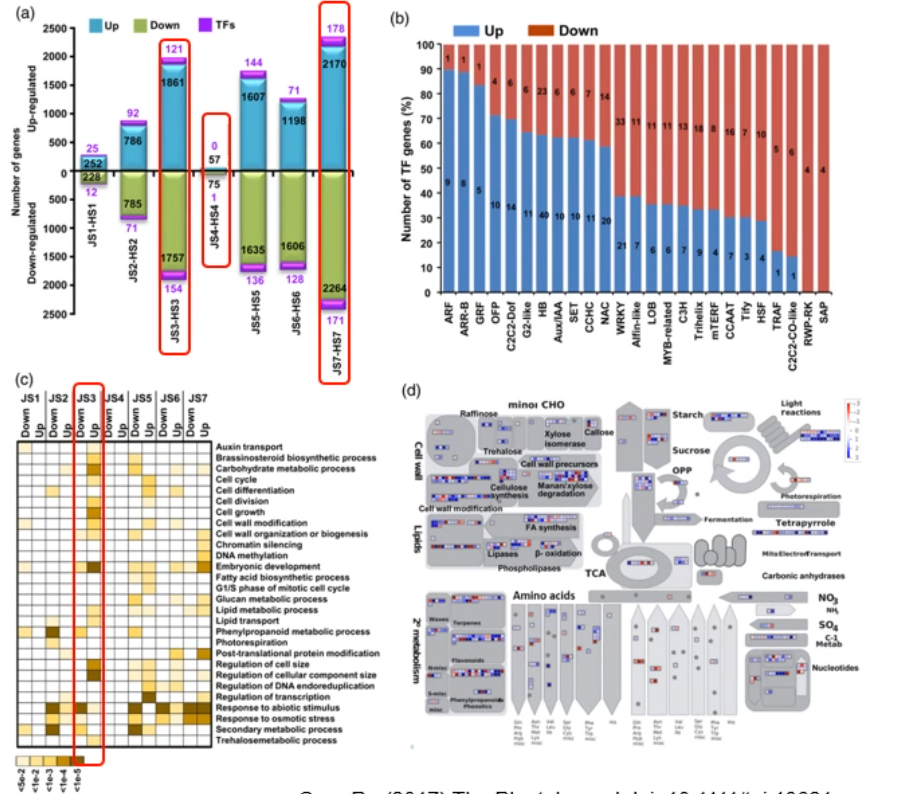
定两个材料在种子发育每个时期的显著差异表达基因集。
HC1 VS JGK3, 共有 8562 个基因上调表达,9023 个下调表达。
差异基因数目最多的是 S7,其次为 S3;最少的为 S4。
重点分析了 TF,许多 TF 家族在 JGK3 中显著上调或下调模式。
GO 富集显示,在 JGK3 中上调表达基因主要富集在一些细胞分裂相关 term 中,尤其在 S3 中。
代谢通路注释分析显示,在 S3 时期某些代谢通路呈现显著的差异。
在 JGK3 中淀粉代谢和光合作用相关基因激活表达,细胞周期和细胞分裂相关基因也上调表达。
在 S3 时期 JGK3 中细胞壁合成和修饰的许多基因上调表达。
# 4.6 WGCNA 鉴定共表达基因模块
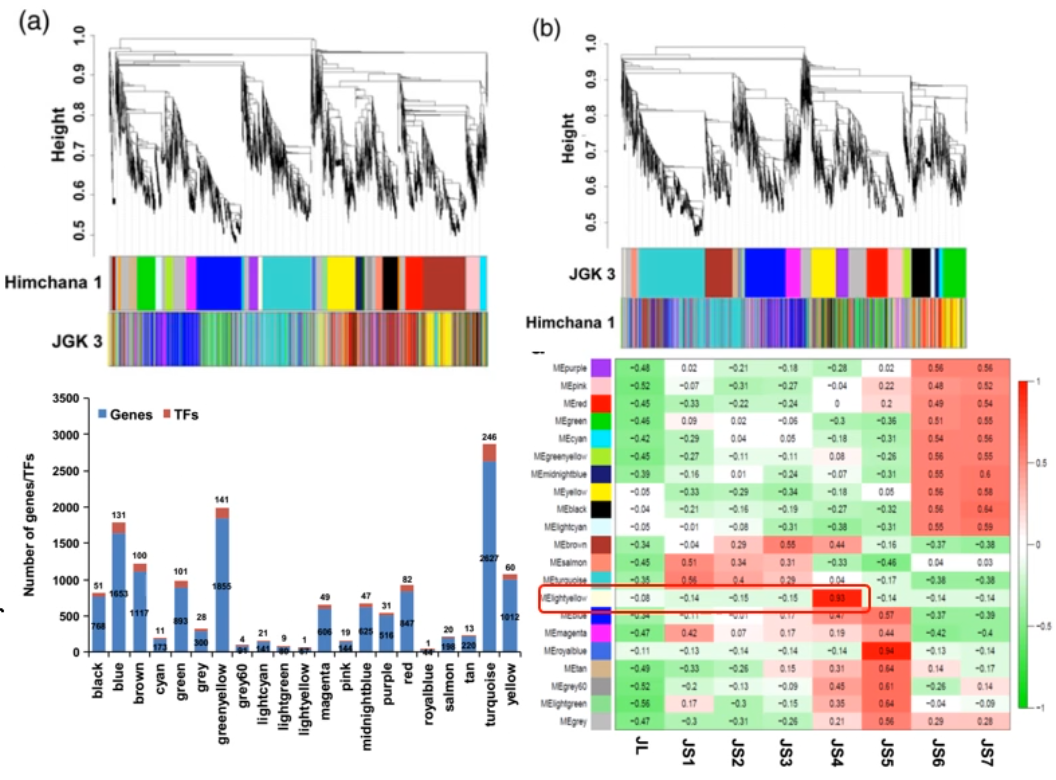
WGCNA 分别鉴定了 HC1 的 27 个基因模块和 JGK3 的 21 个基因模块。
所有模块中都包含 TF,数量从几个到几百个不等。
模块和发育时期样品关联分析(PCC),13 个 HC1 模块和 6 个 JGK3 模块与发育时期样品高度关联(0.6 以上)。
许多模块不仅与一个发育时期关联,一些模块仅与某个特定发育时期样品关联。如 JGK 3 的 lightyellow 模块与 S4 高度特异关联(0.93).
模块的 GO 富集分析结果与差异表达基因分析结果相一致。如,种子发育早期相关模块主要富集的 GO term 为细胞分离、细胞形态、细胞壁组装、基因表达调控。
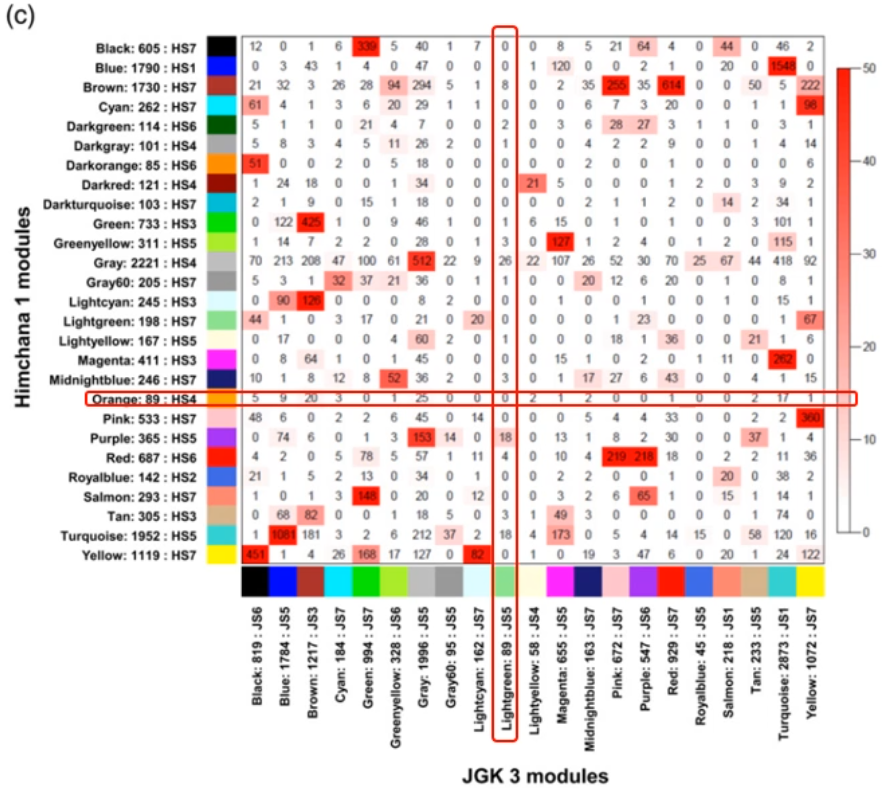
# 4.7 两个材料的基因模块保守性分析
鉴定两个材料共表达基因模块的保守性。
计算不同模块中的相同基因数目,然后通过 Fisher 精确检验的 P-value 值评估显著性。
两个材料中大部分保守模块关联的是相似的种子发育时期样品。
少部分保守模块在不同材料中表型不同的发育时期关联性和转录激活时期。
鉴定了材料特异性模块,如 HC1 的 3 个模块(organe-HS4 等)和 4 个 JGK3 模块(如 lightgreen-JS4)。
HC1 特异性模块主要富集 GO term 为转录调控、细胞程序性死亡、衰老等;JGK3 特异性模块富集的为 DNA 复制、细胞分裂、基因表达、蛋白修饰等。
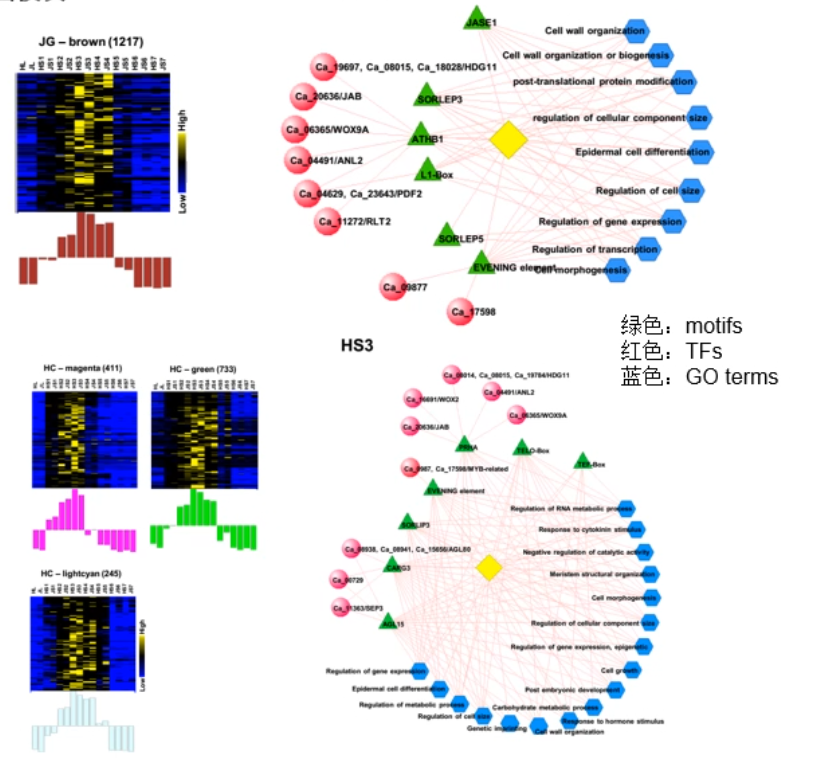
# 4.8 种子发育和种子大小、重量相关转录调控模块分析
目的:鉴定 JGK3 发育 S3 和 S5 的转录调控网络。主要为 TFs 及其共表达的靶基因(包含 TFs 结合位点,motif 显著富集分析)
候选模块:HC1 和 JGK3 中与 S3、S5 时期相关的共表达基因模块
JGK3 的 S3 时期相关模块 brown 转录调控网络:显著富集的 DNA motifs 有 ATHB1、JASE1 等,相关的 TFs 有 woX9、PDF2、RLT2 等,以及靶基因相关的 GO term,基因表达调控、细胞壁组装、表达大小调控等。
比较 JS3 和 HS3 模块转录调控网络,大部分组分是相同的,但是也有一些材料特异性的组分。
同样也分析了 JS5 和 HS5 相关模块转录调控网络组分,包括 DNA motifs、TFs,以及 GO term。
JS5 和 HS5 的调控网络大部分组分是相同的,但是也有一些材料特异性的组分。
该分析鉴定了种子发育中的关键调控因子,两个材料的调控相似但不完全一样。
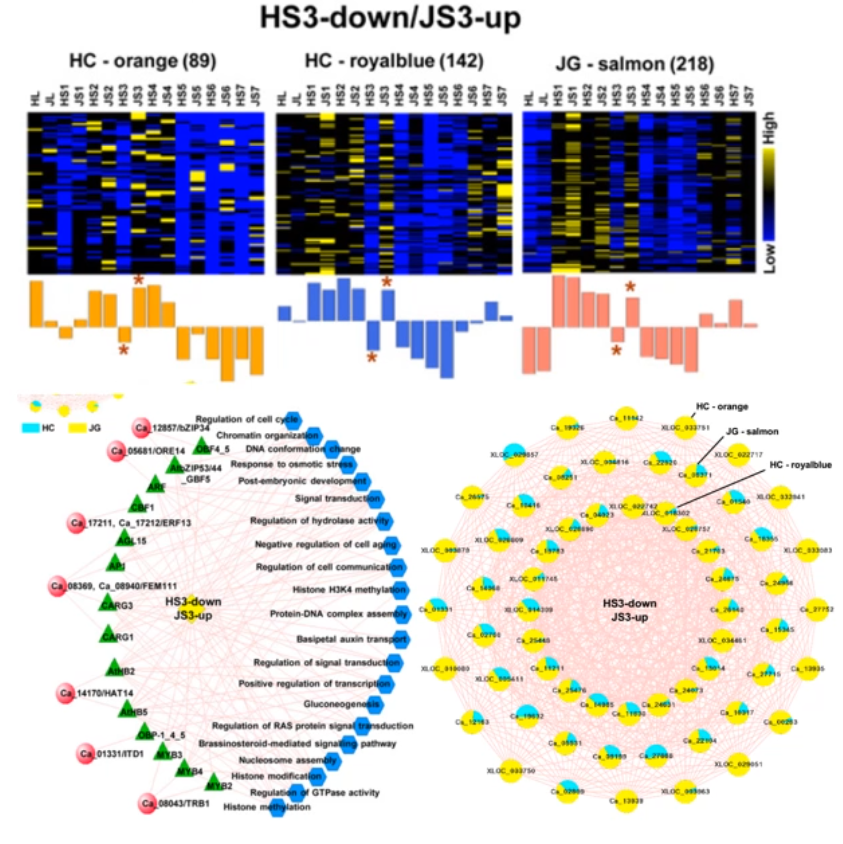
# 4.9 种子发育和种子大小、重量相关转录调控模块分析
一些基因模块在两个材料的 S3 和 S5 时期表现出相反表达模式。
主要有 3 类:HS3 下调 JS3 上调,HS3 上调 JS3 下调,HS5 下调 JS5 上调。
这些模块可能与两个材料种子发育不同相关,进行转录调控网络分析。
HJ3 上调 JS3 下调转录调控网络鉴定:motifs、TF、GOterms。
S3 时期的 top hub 基因表达模式反应了这不同模块中所有基因的表达模式。
这些网络中的许多 motifs、TFs 都是已知参与调控种子大小、重量的重要调控因子。
# 4.10 小结
实验方案:两个材料,不同发育时期样品,所有差异表达基因进行 WGCNA 分析。
两个材料分别进行 WGCNA 分析鉴定各自的基因模块。
通过模块 Epigengene 值与不同发育时期样品进行关联分析,鉴定时期特异性模块,并通过模块 GO 功能富集来解析各发育时期的调控机制。
两个材料模块保守性分析,鉴定保守性和特异性模块,通过 Go 富集解析各自表型。
模块筛选:
依据前面研究结果 S3 和 S5 两个材料差异最大,重点分析这两个时期相关的基因模块。
依据表达模式筛选在两个材料的 S3 和 S5 时期表现出相反表达模式的模块。
转录调控网络关键基因筛选:TFs 和包含显著富集 motifs 的靶基因、top 20/40 hub gene。
# 5. 表型数据
Root Cell-Specific Regulators of Phosphate-Dependent Growth
# 5.1 PRCE 在根部的细胞特异性表达验证和 T-DNA 插入突变体筛选
构建了 12 个 PRCE 基因的启动子 - GFP 转基因 line,验证它们是否呈现细胞特异性表达模式。
其中 10 个基因表现出严格的细胞类型特异性表达模式(皮层、中柱鞘、中柱、木质部薄壁细胞等)。
筛选鉴定了 11 个 PRCE 基因的 T-DNA 插入纯合突变体,其中 10 个为功能缺失突变体,1 个为功能获得型突变体。
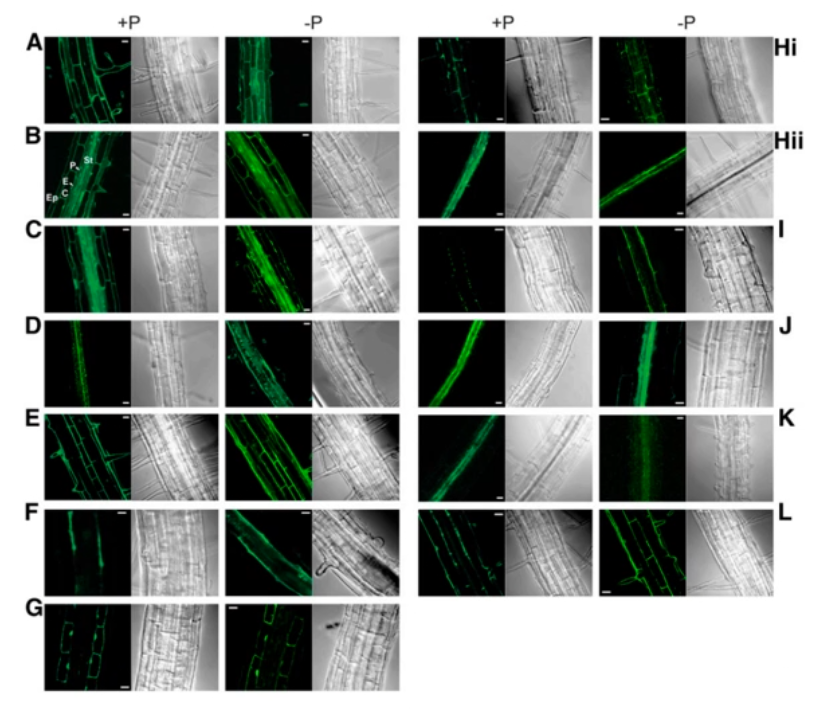
# 5.2 突变体表型分析
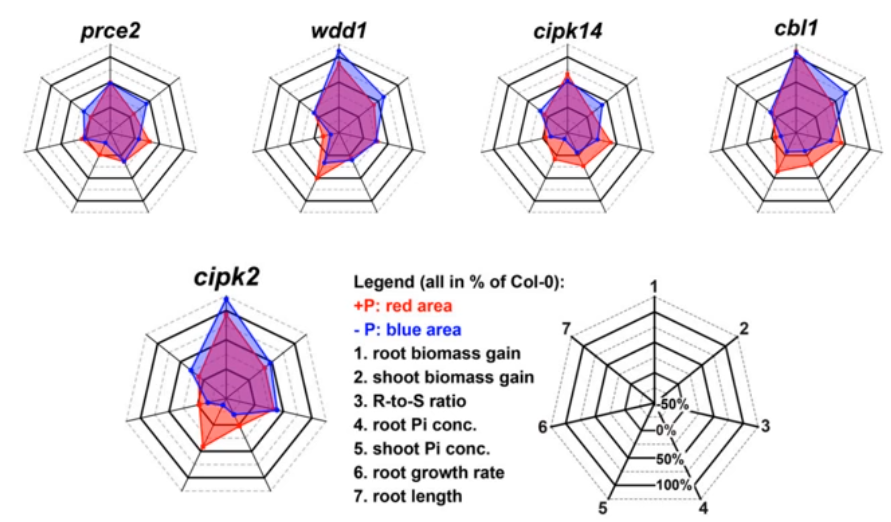
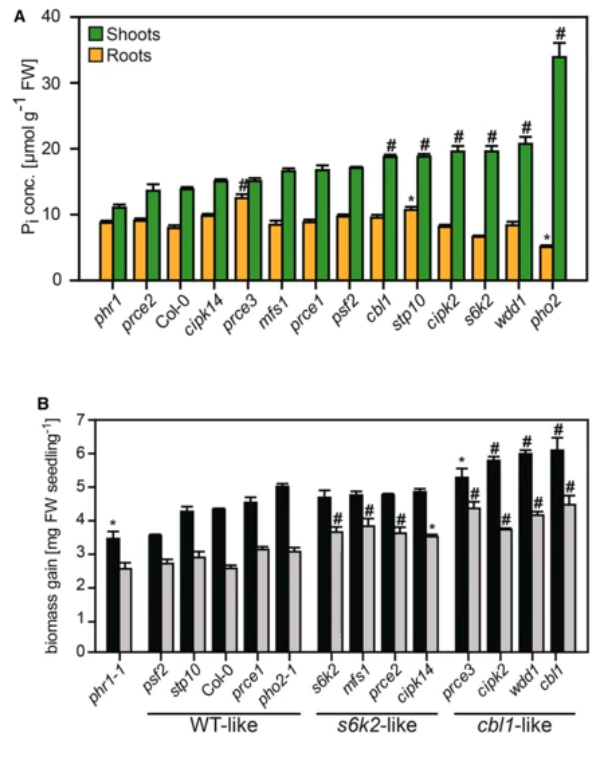
prce 突变体在磷足够和缺乏条件下,植物根和芽中磷的浓度变化。
prce 突变体在磷足够和缺乏条件下,植物生长情况,根和芽中生物量的变化。
大部分 prce 突变体表现出明显不同于野生型(Col-0) 的特征,包括所有定量的生理表型。
# 5.3 prce 突变体根中相应基因的转录水平变化
选取以前发表文献中的缺磷的两个转录组数据集,包含不同的基因型材料,其中 Col-0 和 phr1-1 为对照材料。
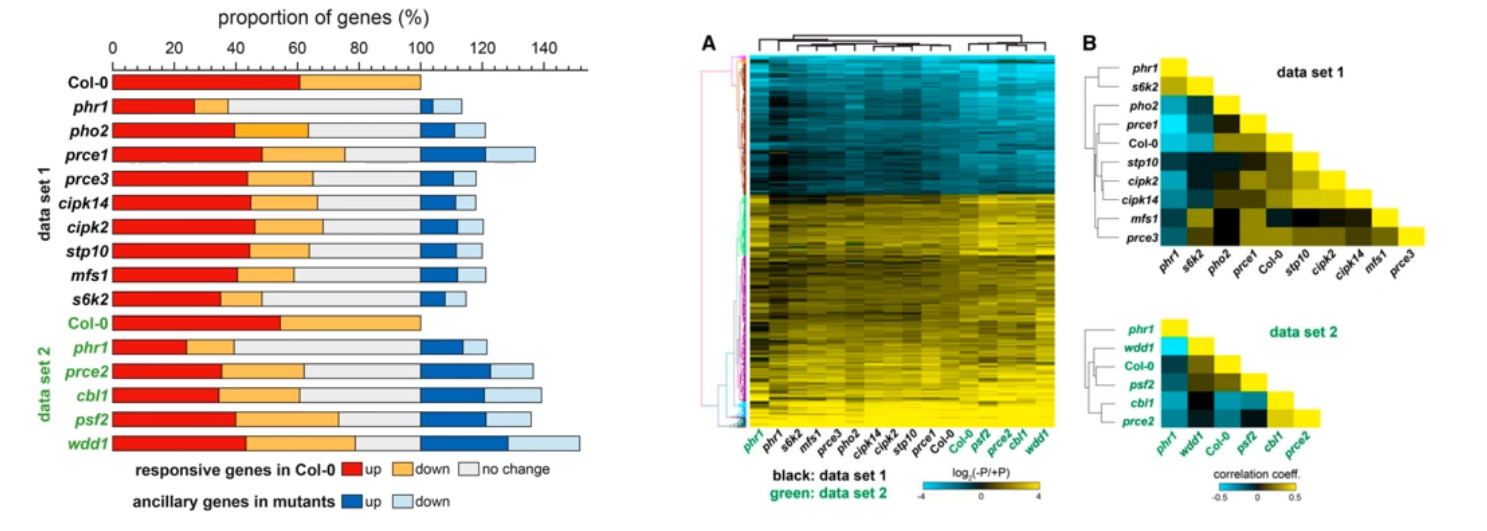
在两个数据集中,Col-0 的 63% 和 6% 的 PSR 基因在 phr1-1 中没有变化;许多野生型 PSR 基因在 prce 突变体中呈现出不同的表达。
在两个数据集中筛选了 Col-0 中差异表达 2 倍以上的 831 个磷响应核心基因,进步通过层次聚类分析其在不同基因型材料中的表达模式。并依据基因表达模式分析不同基因型样品之间的关系。
S6k2 突变体表现出与 phr1-1 类似的缺磷反应,而 wdd1 突变体表现出类似 Col-0 的缺磷反应。
# 5.4 鉴定 prce 突变体相关共表达网络
对 32 个 RNA-seq 数据集(磷足够和磷缺乏)的所有表达转录本分别进行 WGCNA 分析,都鉴定了 18 个共表达基因模块。
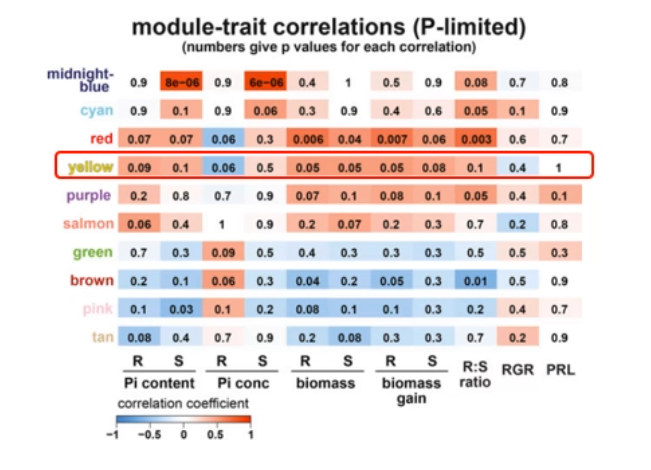
计算模块的特征值(Eigengene),并通过特征值来计算模块和生理性状(数量性状,如磷含量和浓度、生物量、根相对生长速率、初根根长等) 的相关性。
重点关注与性状显著相关的 10 个模块,以及在不同基因型中呈现相反表达模式的模块。
缺磷时,yellowf 和 red 模块与生物量显著正相关;磷充足时,black 模块与生物量显著负相关。
与生物量呈现相反关联的还有缺磷的 green 模块和磷足够的 pink 模块。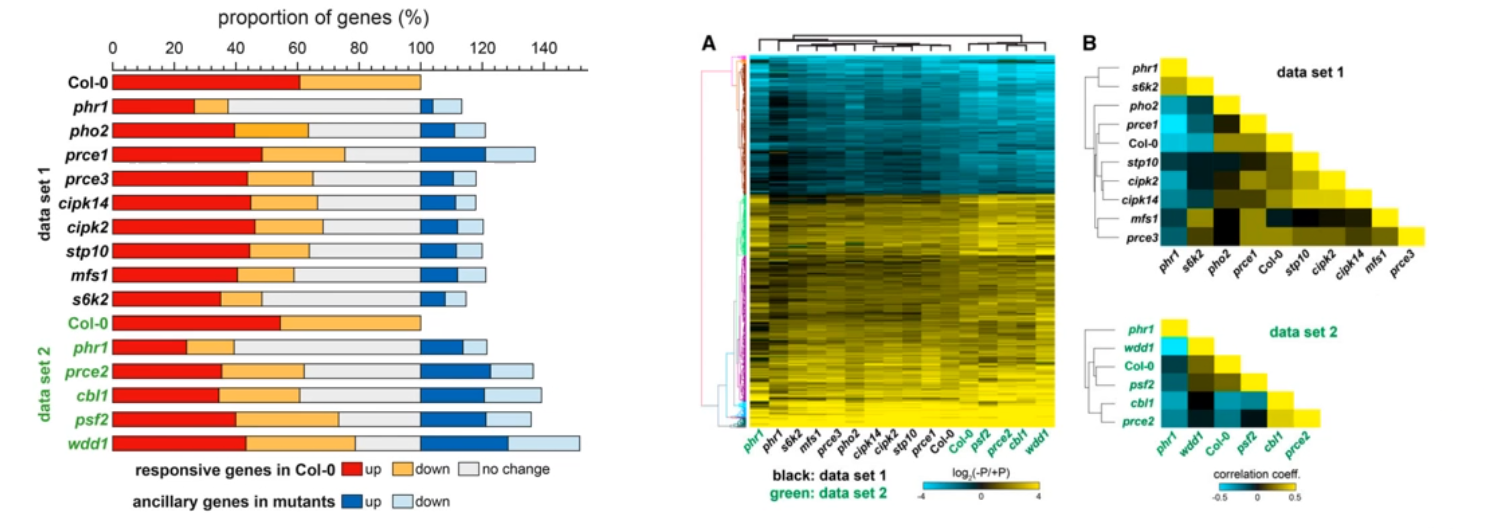
# 5.5 重点模块和模块内 hub 基因分析
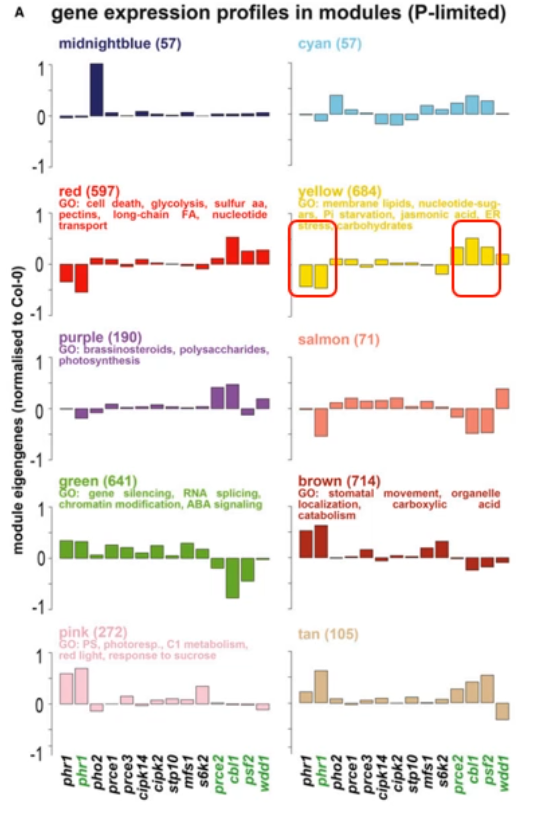
缺磷的 yellow 模块,包含 684 个基因,与生物量、磷含量、根芽比例都显著相关。其中 24% 基因与之前转录组鉴定的 PSI 基因相一致。
模块基因,相对野生型,在 phr1-1 中下调表达,在 prce 突变体(cb/1、prce2 等)上调表达。
筛选与该模块的 ME(kME) 排在前 300 的基因进行富集分析,显著富集的 GO term 有缺磷相关、磷脂和半乳糖脂代谢等。
模块 hub gene 筛选:kME 大于 0.9。主要为脂代谢、感知磷、磷信号导、磷运输等相关基因。
Yellow 模块在 cb/1 中表现较高的 ME 值,说明钙信号通过 CBL1 影响磷转运。进一步筛选该模块中钙信号相关基因,重点关注,作为 hub gene 候选。
# 5.6 小结
实验方案:两种处理,不同基因型样品,所有表达基因进行 WGCNA 分析。
两种处理分别进行 WGCNA 分析鉴定各自的基因模块。
模块筛选:
通过模块 Epigengene 值与不同表型(数量性状)进行关联分析,筛选性状相关模块;筛选在缺磷和磷足够条件下与表型呈现相反关联的模块。
模块功能分析:GO 功能富集分析。
模块 hub gene 筛选:
与模块的 kME 值大于 0.9;分析模块特征值在各基因型样品中表达模式,筛选关联高的突变体,重点关注突变基因及相关通路基因。
